번역 후 변형
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
번역 후 변형은 단백질이 번역된 후 발생하는 화학적 변형을 의미하며, 단백질의 기능, 활성, 위치, 상호작용 등을 조절하는 중요한 과정이다. 번역 후 변형에는 작용기 추가, 다른 단백질 또는 펩타이드와의 결합, 아미노산의 화학적 변형, 구조적 변형 등 다양한 종류가 있으며, 인산화, 아세틸화, 글리코실화 등이 흔하게 발생한다. 이러한 변형은 효소에 의해 조절되거나, 비효소적으로 발생하기도 하며, 단백질 서열을 기반으로 예측하거나, 데이터베이스를 통해 확인할 수 있다. 관련 데이터베이스로는 프로테옴스카우트, 인간 단백질 참조 데이터베이스, PROSITE 등이 있으며, PyMOL, AWESOME, 키메라와 같은 도구들을 활용하여 연구할 수 있다.
더 읽어볼만한 페이지
- 단백질 생합성 - 리보솜
리보솜은 rRNA와 단백질로 구성된 두 개의 소단위체로 이루어진 세포 내 단백질 합성 복합체로서, 세포질 내에서 mRNA의 코돈을 해독하여 폴리펩타이드 사슬을 합성하는 번역 과정을 수행하며, 원핵세포와 진핵세포의 구조적 차이로 인해 항생제 개발의 표적이 되고, RNA 세계에서 기원한 자기 복제 복합체로 추정된다. - 단백질 생합성 - 종결 코돈
종결 코돈은 표준 유전 암호에서 단백질 합성을 종료시키는 역할을 하는 UAG(앰버), UAA(오커), UGA(오팔 또는 엄버) 세 가지 코돈이며, 넌센스 돌연변이, 논스톱 돌연변이 등 다양한 유전 질환과 관련이 있을 수 있다. - 번역 후 변형 - 단백질 카보닐화
- 번역 후 변형 - 메틸화
메틸화는 메틸기를 분자에 첨가하는 과정으로, 생물학에서 유전자 발현 조절 등 생명현상에 관여하고 유기화학에서 유기 합성에 활용되며, 다양한 생물종에 존재하는 중요한 생리적 과정이다. - 단백질 구조 - 메틸화
메틸화는 메틸기를 분자에 첨가하는 과정으로, 생물학에서 유전자 발현 조절 등 생명현상에 관여하고 유기화학에서 유기 합성에 활용되며, 다양한 생물종에 존재하는 중요한 생리적 과정이다. - 단백질 구조 - 녹색 형광 단백질
녹색 형광 단백질(GFP)은 해파리에서 발견된 단백질로, 특정 파장의 빛을 흡수하여 녹색 형광을 방출하며, 자체 발색단 형성 능력과 유전자 조작을 통한 개량형 개발로 생물학 연구에서 리포터 유전자로 널리 사용되지만, 실험 결과 해석 시 주의가 필요하고 대체 기술 또한 개발 중이다.
2. 번역 후 변형의 종류
단백질 합성이 완료된 후 일어나는 번역 후 변형(PTM)은 그 종류가 매우 다양하며, 변형 방식이나 결과에 따라 여러 기준으로 분류할 수 있다. 주요 분류 기준은 다음과 같다.
- '''작용기 추가''': 인산화, 아세틸화, 메틸화, 글리코실화 등 특정 화학 작용기가 아미노산 잔기에 공유 결합으로 부가되는 변형이다. 이는 단백질의 활성, 안정성, 상호작용 등을 조절하는 데 중요한 역할을 한다.
- '''다른 단백질 또는 펩타이드와의 결합''': 유비퀴틴이나 SUMO 단백질과 같은 다른 단백질 또는 펩타이드가 표적 단백질에 결합하는 변형이다. 유비퀴틴화, SUMO화 등이 대표적이며, 주로 단백질 분해 조절이나 신호 전달 경로에 관여한다.
- '''아미노산의 화학적 변형''': 단백질을 구성하는 아미노산 자체의 화학 구조가 변화하는 변형이다. 예를 들어 아르기닌이 시트룰린으로 바뀌는 시트룰린화나 아스파라긴이 아스파르트산으로 바뀌는 탈아미드화 등이 있다.
- '''구조적 변형''': 단백질의 전체적인 구조에 영향을 미치는 변형이다. 시스테인 잔기 사이에 형성되는 이황화 결합, 특정 부위가 잘려나가는 단백질 분해 절단, 단백질의 일부가 제거되고 남은 부분이 연결되는 단백질 스플라이싱 등이 여기에 속한다.
이러한 다양한 변형의 구체적인 종류와 기능에 대해서는 아래 하위 문단에서 자세히 설명한다.
2. 1. 작용기 추가
단백질의 특정 아미노산 잔기에 다양한 화학 작용기가 추가되는 변형을 의미한다. 이러한 변형은 단백질의 기능, 안정성, 세포 내 위치 등을 조절하는 중요한 메커니즘이다. 주요 작용기 추가 변형으로는 아실화, 알킬화, 글리코실화, 인산화, 리포일화, 하이드록실화 등이 있다. 이러한 변형들은 효소의 도움을 받아 일어나기도 하고(효소적 변형), 효소 없이 자발적으로 발생하기도 한다(비효소적 변형). 각 변형의 구체적인 종류와 메커니즘, 생물학적 중요성에 대한 자세한 내용은 아래 하위 문단에서 설명한다.2. 1. 1. 효소에 의한 작용기 추가 (''in vivo'')

아세틸기 부가로, 라이신 잔기 또는 N 말단 아미노기에 발생한다. 에타노일화라고도 한다. 히스톤이 잘 알려진 예이다. 히스톤 꼬리 부분의 아세틸화, 또한 그 역반응인 탈아세틸화는 라이신이 가진 전하를 변화시켜 DNA와의 결합 상태에 영향을 미치는 것과 동시에, 브로모도메인을 가진 히스톤 변형 효소나 염색질 리모델링 인자 등에 대한 표적 부위를 규정하는 히스톤 코드의 요소로서 염색질 구조의 변환에 기여하여 유전자 발현의 변화를 일으킨다.
- **포르밀화
- 알킬화
- * 메틸기 부가는 메틸화라고 하며, 라이신과 아르기닌 잔기에 발생한다.
- * 디메틸화
- 아미드화는 C 말단에서 일어난다.
- 비오티닐화
- * 비오틴을 부가하여 라이신 잔기를 보호한다.
- γ-카복실화
- * 비타민 K에 의존한다.[40]
- 글루타밀화
- * 단백질의 글루탐산 잔기의 공유 결합에 의한 튜불린과 다른 단백질과의 결합[41]
- 글리코실화
- * 아스파라긴, 하이드록시라이신, 세린, 트레오닌에 글리코실기가 부가되어 당단백질이 생성된다. 마이야르 반응에서는 당 부가는 효소 없이 행해진다.
- 글리실화
- * 튜불린의 C 말단 꼬리의 40개 이상의 글리신 잔기 중 하나에 공유 결합된다.
- 헴
- * 공유 결합의 부가
- 하이드록실화
- 요오드화
- * 갑상선 호르몬
- 이소프레닐화
- * 이소프레노이드 부가(파르네솔, 게라닐게라니올 등)
- 리포일화
- * 프레닐화
- * GPI 앵커 형성
- ** 미리스토일화
- ** 파르네실화
- ** 게라닐게라닐화
- 뉴클레오티드 또는 유도체에 공유 결합 부가
- * ADP 리보실화
- * FAD 결합
- * 아데닐릴화
- 산화 환원 반응
- 폴리에틸렌 글리콜화
- 포스파티딜이노시톨
- 포스포판테테이닐화
- * 지방산, 폴리케타이드, 비리보솜 펩티드, 로이신에서 조효소 A로부터 4'-포스포판테테이닐 부가.
- 인산화
- * 인산 부가. 일반적으로 세린, 티로신, 트레오닌, 히스티딘에서 일어난다.
- 피로글루탐산 형성
- 라세미화
- * 프롤린 이성질체화 효소에 의한 프롤린 라세미화
- tRNA
- * 아미노산 부가에 의한 조절
- 티로신 황산화
- * 티로신에 황 부가
- 셀레노일화
- * 셀레노단백질에 셀렌 부가
2. 1. 2. 비효소적 변형 (''in vivo'')
비효소적 PTM은 효소의 도움 없이 자발적으로 일어나는 변형으로, 당화, 당산화, 니트로실화, 산화, 숙신산화 및 지질 과산화 등이 있다.[15] 구체적인 예시는 다음과 같다.- 당화: 효소의 조절 작용 없이 단백질에 당 분자가 첨가되는 현상.
- 카바모일화: 단백질의 N-말단 또는 라이신의 곁사슬에 아이소사이안산이 첨가되는 현상.[16]
- 카보닐화: 일산화 탄소가 다른 유기 또는 무기 화합물에 첨가되는 현상.
- 자발적인 아이소펩타이드 결합 형성: 그람 양성 세균의 많은 표면 단백질에서 발견된다.[17]
2. 1. 3. 비효소적 변형 (''in vitro'')
- 바이오티닐화: 바이오틴 분자를 시약을 이용해 공유 결합시키는 변형으로, 주로 단백질 표지에 사용된다.
- 카바모일화: 이소시안산이 단백질의 N-말단 또는 라이신(Lys)이나 시스테인(Cys) 잔기의 곁사슬에 첨가되는 변형이다. 일반적으로 요소 용액에 노출될 때 발생한다.[18]
- 산화: 하나 이상의 산소 원자가 반응하기 쉬운 곁사슬, 주로 메티오닌(Met), 트립토판(Trp), 히스티딘(His) 또는 시스테인(Cys) 잔기에 첨가되는 변형이다. 시스테인 잔기 사이의 이황화 결합 형성을 포함한다.
- 페길화: 시약을 사용하여 폴리에틸렌 글리콜(PEG) 분자를 공유 결합시키는 변형으로, 주로 단백질의 N-말단 또는 라이신(Lys) 잔기의 곁사슬에 적용된다. 페길화는 단백질 의약품의 효능을 향상시키는 데 사용된다.
2. 2. 다른 단백질 또는 펩타이드와의 결합
- 유비퀴틴화: 단백질 유비퀴틴과의 공유 결합 연결.
- SUMO화: SUMO 단백질(Small Ubiquitin-related MOdifier, 작은 유비퀴틴 유사 변형 인자)과의 공유 결합 연결.[19]
- NEDD화: Nedd 단백질과의 공유 결합 연결.
- ISG화: ISG15 단백질(Interferon-Stimulated Gene 15, 인터페론 자극 유전자 15)과의 공유 결합 연결.[20]
- Pup화: 원핵생물 유비퀴틴 유사 단백질과의 공유 결합 연결.
- 글루타밀화: 단백질의 글루탐산 잔기의 공유 결합을 통해 튜불린과 다른 단백질이 결합하는 변형.[41]
- 글리실화: 튜불린의 C 말단 꼬리에 있는 40개 이상의 글리신 잔기 중 하나에 다른 분자가 공유 결합하는 변형.
- tRNA 매개 아미노산 부가: 아미노산 부가를 통한 조절.
2. 3. 아미노산의 화학적 변형
아미노산의 화학적 구조가 변형되는 주요 사례는 다음과 같다.
- '''시트룰린화''' 또는 '''데이민화''': 아르기닌이 시트룰린으로 전환되는 반응[21]
- '''탈아미드화''': 글루타민이 글루탐산으로, 또는 아스파라긴이 아스파르트산으로 전환되는 반응
- '''엘리미닐화''': 인산트레오닌 및 인산세린의 베타-제거 또는 탈수를 통해 알켄으로 전환되는 반응. 트레오닌 및 세린에서 일어난다.[22]
- '''아실화'''
- 아실기(RCO-) 부가. ''O''-아실화(에스터화), ''N''-아실화(아미드화), ''S''-아실화(티오에스터화) 등이 있다.
- '''아세틸화''': 아세틸기 부가 반응으로, 라이신 잔기 또는 N 말단 아미노기에서 발생한다. 에타노일화라고도 한다. 히스톤의 아세틸화가 대표적인 예이다. 히스톤 꼬리 부분의 아세틸화 및 탈아세틸화는 라이신의 전하를 변화시켜 DNA와의 결합에 영향을 준다. 또한, 브로모도메인을 가진 히스톤 변형 효소나 염색질 리모델링 인자 등의 표적 부위를 제공하는 히스톤 코드의 요소로서 염색질 구조 변환 및 유전자 발현 조절에 기여한다.
- '''포르밀화'''
- '''알킬화'''
- 메틸기 부가 ('''메틸화'''): 라이신과 아르기닌 잔기에서 발생한다.
- '''디메틸화'''
- '''아미드화''': C 말단에서 일어난다.
- '''비오티닐화''': 비오틴을 부가하여 라이신 잔기를 보호한다.
- '''γ-카복실화''': 비타민 K 의존적인 반응이다.[40]
- '''글루타밀화''': 단백질의 글루탐산 잔기에 공유 결합을 통해 튜불린과 다른 단백질을 연결한다.[41]
- '''글리코실화''': 아스파라긴, 하이드록시라이신, 세린, 트레오닌에 글리코실기가 부가되어 당단백질이 생성된다. 마이야르 반응에서는 효소 없이 당 부가가 일어난다.
- '''글리실화''': 튜불린의 C 말단 꼬리에 있는 40개 이상의 글리신 잔기 중 하나에 공유 결합으로 연결된다.
- '''헴''': 공유 결합으로 부가된다.
- '''하이드록실화'''
- '''요오드화''': 갑상선 호르몬 생성 과정에서 일어난다.
- '''이소프레닐화''': 이소프레노이드(파르네솔, 게라닐게라니올 등)가 부가된다.
- '''리포일화'''
- '''프레닐화'''
- '''GPI 앵커 형성'''
- '''미리스토일화'''
- '''파르네실화'''
- '''게라닐게라닐화'''
- '''뉴클레오티드 또는 유도체 부가''': 공유 결합으로 부가된다.
- '''ADP 리보실화'''
- '''FAD 결합'''
- '''아데닐릴화'''
- '''산화 환원 반응'''
- '''폴리에틸렌 글리콜화'''
- '''포스파티딜이노시톨'''
- '''포스포판테테이닐화''': 지방산, 폴리케타이드, 비리보솜 펩티드, 로이신 등에서 조효소 A로부터 4'-포스포판테테이닐기가 부가된다.
- '''인산화''': 인산기가 부가되는 반응으로, 주로 세린, 티로신, 트레오닌, 히스티딘에서 일어난다.
- '''피로글루탐산 형성'''
- '''라세미화'''
- 프롤린 이성질체화 효소에 의한 프롤린의 라세미화
- '''tRNA''': 아미노산 부가에 의한 조절
- '''티로신 황산화''': 티로신에 황이 부가된다.
- '''셀레노일화''': 셀레노단백질에 셀렌이 부가된다.
2. 4. 구조적 변형
- 이황화 결합: 두 개의 시스테인 아미노산 잔기가 공유 결합을 형성하는 것.
- 라이신-시스테인 결합: 산소 원자를 매개로 하나의 라이신과 하나 또는 두 개의 시스테인 잔기가 공유 결합하는 것 (NOS 및 SONOS 결합).[23]
- 단백질 분해 절단: 프로테아제(단백질 분해 효소)에 의해 단백질의 펩타이드 결합이 가수 분해되어 절단되는 과정.
- 아이소아스파르트산 형성: 아스파라긴 또는 아스파트산 아미노산 잔기가 고리화 반응을 일으키는 것.
- 래스화 (Racemization): 아미노산의 카이랄성 중심 입체 배열이 바뀌는 현상.
- * 단백질-세린 에피머라제에 의한 세린의 래스화.
- * 개구리 오피오이드 펩타이드인 데르모르핀에 포함된 알라닌의 래스화.
- * 개구리 오피오이드 펩타이드인 델토르핀에 포함된 메티오닌의 래스화.
- 단백질 스플라이싱: 단백질의 특정 부분(인테인)이 스스로 잘려나가고 남은 부분(엑스테인)이 다시 연결되는 과정으로, mRNA 스플라이싱과 유사한 자가 촉매 반응이다.
3. 번역 후 변형의 통계
번역 후 변형은 다양한 단백질의 기능 조절에 필수적이며, 어떤 종류의 변형이 얼마나 자주 발생하는지에 대한 통계적 연구가 이루어지고 있다. 예를 들어, 인슐린 형성 과정에서는 가수분해와 이황화물 형성이 중요하며, 히스톤 단백질의 변형은 크로마틴 구조와 RNA 중합 효소의 접근성을 조절하여 유전자 발현을 제어하는 데 핵심적인 역할을 한다. 또한 RNA 중합 효소 II 자체의 변형도 전사 과정을 조절하는 중요한 기작이다. 이러한 다양한 변형들의 구체적인 발생 빈도와 아미노산 잔기별 종류에 대한 통계는 아래 하위 섹션에서 자세히 다룬다.
3. 1. 빈도별 흔한 변형
2011년, Swiss-Prot 데이터베이스의 프로테옴 전체 정보를 사용하여 실험적 및 추정적으로 감지된 각 번역 후 변형의 통계가 수집되었다.[24] 이 중 실험적으로 가장 흔하게 발견된 상위 10가지 변형은 다음과 같다.[25]
3. 2. 잔기별 흔한 변형
달리 표시되지 않는 한 변형은 측쇄에서 발생한다.
4. 데이터베이스 및 도구
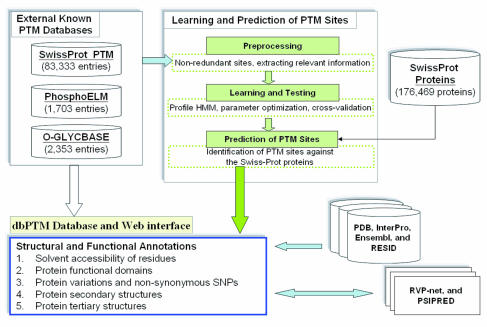
단백질 서열은 변형 효소에 의해 인식되는 서열 모티프를 포함하며, 이는 PTM 데이터베이스에 기록되거나 예측 도구를 통해 예측될 수 있다. 다양한 종류의 변형이 많이 발견되면서, 이러한 정보를 데이터베이스에 체계적으로 정리할 필요성이 커졌다. PTM 정보는 실험을 통해 직접 수집하거나, 신뢰도 높은 데이터를 기반으로 예측하여 얻을 수 있다. 현재 수많은 데이터베이스가 개발되었으며, 이들은 종종 특정 분류군(예: 인간 단백질)이나 다른 특정 특징에 초점을 맞추어 정보를 제공한다.
4. 1. 데이터베이스 목록
- [http://www.phosphosite.org/ PhosphoSitePlus] – 포유류 단백질 번역 후 변형 연구를 위한 포괄적인 정보와 도구를 제공하는 데이터베이스이다.
- 프로테옴스카우트 – 실험적으로 확인된 단백질 및 번역 후 변형에 대한 데이터베이스이다.
- 인간 단백질 참조 데이터베이스 – 다양한 변형에 대한 데이터베이스로, 질병 유발 단백질과 관련된 다양한 단백질, 그 종류, 기능/과정을 이해하는 데 도움을 준다.
- PROSITE – 여러 유형의 PTM(단백질 번역 후 변형)에 대한 컨센서스 패턴 데이터베이스이며, 사이트를 포함한다.
- RESID – PTM에 대한 주석 및 구조 모음을 포함하는 데이터베이스이다.
- [https://research.bioinformatics.udel.edu/iptmnet/ iPTMnet] – 여러 지식 베이스와 텍스트 마이닝 결과를 통합한 PTM 정보 데이터베이스이다.
- dbPTM – 다양한 PTM과 관련된 정보(화학적 구성 요소/구조, 아미노산 변형 부위 빈도 등)를 보여주는 데이터베이스이다.
- [https://www.uniprot.org/help/post-translational_modification Uniprot]은 PTM 정보를 제공하지만, 더 전문적인 데이터베이스보다 덜 포괄적일 수 있다.
- [https://www.oglcnac.mcw.edu/ The ''O''-GlcNAc Database] - 단백질 O-GlcNAc화에 대한 큐레이션된 데이터베이스로, 14,000개 이상의 단백질 항목과 10,000개 이상의 ''O''-GlcNAc 사이트를 참조한다.
4. 2. 도구
- PyMOL[35] – 단백질 모델에 일반적인 PTM 세트를 도입한다.
- AWESOME[36] – 단일 염기 다형성이 PTM에 미치는 역할을 확인하는 대화형 도구이다.
- 키메라[37] – 분자를 시각화하는 대화형 데이터베이스이다.
5. 대표적인 예시
참조
[1]
서적
Fundamentals of Biochemistry: Life at the Molecular Level
https://books.google[...]
Wiley
2006
[2]
논문
Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database
2011-09
[3]
서적
Molecular Cell Biology
https://archive.org/[...]
W. H. Freeman
2000
[4]
논문
Protein carbonylation, cellular dysfunction, and disease progression
[5]
논문
Oxidative stress and covalent modification of protein with bioactive aldehydes
2008-08
[6]
논문
Detecting oxidative post-translational modifications in proteins
2007-07
[7]
서적
Posttranslational modification of proteins : expanding nature's inventory
Roberts and Co. Publ.
2006
[8]
논문
Murine elongation factor 1 alpha (EF-1 alpha) is posttranslationally modified by novel amide-linked ethanolamine-phosphoglycerol moieties. Addition of ethanolamine-phosphoglycerol to specific glutamic acid residues on EF-1 alpha
1989-08
[9]
논문
The tRNA synthetase paralog PoxA modifies elongation factor-P with (R)-β-lysine
2011-08
[10]
논문
Lysine Acetylation Goes Global: From Epigenetics to Metabolism and Therapeutics
2018-02
[11]
논문
Peptide amidation
1991-03
[12]
논문
Posttranslational glutamylation of alpha-tubulin
1990-01
[13]
논문
On a potential global role for vitamin K-dependent gamma-carboxylation in animal systems. Evidence for a gamma-glutamyl carboxylase in Drosophila
2001-03
[14]
논문
Cysteine oxidative posttranslational modifications: emerging regulation in the cardiovascular system
2013-01
[15]
간행물
"The Advanced Lipoxidation End-Product Malondialdehyde-Lysine in Aging and Longevity"
[16]
논문
Carbamylation-derived products: bioactive compounds and potential biomarkers in chronic renal failure and atherosclerosis
2011-11
[17]
논문
Intramolecular isopeptide bonds: protein crosslinks built for stress?
2011-04
[18]
논문
Reactions of the Cyanate Present in Aqueous Urea with Amino Acids and Proteins
[19]
웹사이트
Sumoylation: Molecular Biology and Biochemistry
http://www.horizonpr[...]
2005-02-09
[20]
논문
Protein ISGylation modulates the JAK-STAT signaling pathway
2003-02
[21]
논문
Immunity to citrullinated proteins in rheumatoid arthritis
https://zenodo.org/r[...]
[22]
논문
Eliminylation: a post-translational modification catalyzed by phosphothreonine lyases
2009-03
[23]
논문
Widespread occurrence of covalent lysine–cysteine redox switches in proteins
2022-04
[24]
논문
Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database
2011-09
[25]
웹사이트
Proteome-Wide Post-Translational Modification Statistics
http://selene.prince[...]
2011-07-22
[26]
논문
dbPTM: an information repository of protein post-translational modification
2006-01
[27]
논문
PhosphoSitePlus, 2014: mutations, PTMs and recalibrations
2015-01
[28]
논문
Human Protein Reference Database and Human Proteinpedia as resources for phosphoproteome analysis
2012-02
[29]
논문
PROSITE, a protein domain database for functional characterization and annotation
2010-01
[30]
논문
The RESID Database of Protein Modifications: 2003 developments
2003-01
[31]
논문
iPTMnet: an integrated resource for protein post-translational modification network discovery
2018-01
[32]
논문
In silico prediction tools and molecular modeling
2017-03-31
[33]
논문
The human ''O''-GlcNAcome database and meta-analysis
2021-01
[34]
논문
Automatization and self-maintenance of the O-GlcNAcome catalog: a smart scientific database
2021-07
[35]
논문
PyTMs: a useful PyMOL plugin for modeling common post-translational modifications
2014-11
[36]
논문
AWESOME: a database of SNPs that affect protein post-translational modifications
2019-01
[37]
논문
structureViz: linking Cytoscape and UCSF Chimera
2007-09
[38]
웹사이트
1tp8 - Proteopedia, life in 3D
http://www.proteoped[...]
[39]
간행물
Abgent Catalog
2004-05
[40]
웹사이트
http://www.jbc.org/c[...]
2001
[41]
웹사이트
http://www.sciencema[...]
[42]
논문
Protein ISGylation modulates the JAK-STAT signaling pathway
http://www.genesdev.[...]
2003
[43]
서적
Sumoylation: Molecular Biology and Biochemistry
http://www.horizonpr[...]
Horizon Bioscience
2004
[44]
서적
Fundamentals of biochemistry : life at the molecular level
https://archive.org/[...]
Wiley
2006
[45]
저널
Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database
2011-09
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com